Taula de continguts:
- Pas 1: organització de les dades de mamografia
- Pas 2: processament d'imatges
- Pas 3: Codi de llindar
- Pas 4: trobar anomalies per a cada imatge binària
- Pas 5: representació de la ubicació i la mida de la massa diagnosticada per a la comparació visual
- Pas 6: Implementació del segon mètode de comparació
- Pas 7: analitzar les dades recollides
- Pas 8: Feu el vostre propi classificador
- Pas 9: millores? Alguna idea?
- Autora John Day day@howwhatproduce.com.
- Public 2024-01-30 08:14.
- Última modificació 2025-01-23 14:38.
L’objectiu d’aquest projecte era identificar i utilitzar un paràmetre per processar imatges de mamografia en escala de grisos de diverses classificacions de teixits de fons: teixit gras, greix glandular i dens. Aquesta classificació s’utilitza quan els radiòlegs analitzen mamografies i han de tenir en compte si la densitat de teixits ocultarà alguna anomalia, com ara lesions o tumors. Això es deu al fet que tant les estructures fisiològiques normals com el teixit glandular com el teixit connectiu fibrós. i morfologies anormals com calcificacions i tumors apareixeran molt brillants a la mamografia, mentre que els teixits grassos menys densos apareixeran negres. Per tant, era apropiat programar un classificador que pugui manipular els nivells d’intensitat de píxels per visualitzar i identificar les masses de la millor manera possible.
Pas 1: organització de les dades de mamografia
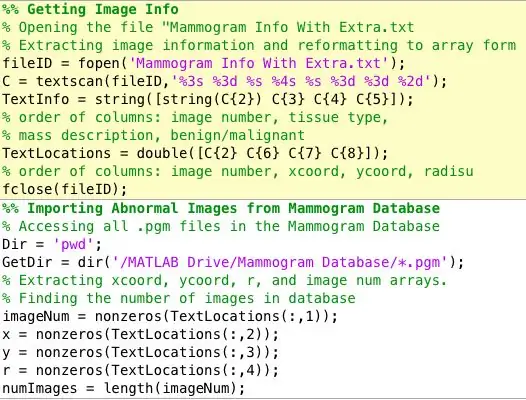
Una de les primeres coses que em vaig adonar que havia de manejar va ser organitzar les dades d’una manera molt clara, concisa i accessible. Aquestes són les variables que he extret de la base de dades de mamografies mini-MIAS. Vaig crear dues matrius. Una que conté 4 columnes:
- Número de la imatge:
- coordenada x de massa
- y coordenada de massa
- Radi de massa: (Definia una mida aproximada per a la massa
La segona matriu contenia informació de classificació:
- Tipus de teixit de fons: greix (F), greix glandular (G), dens (D)
- Descripció de la massa: ben definida (CIRC), espiculada (SPIC), mal definida (MISC) Distorsió arquitectònica (ARCH), asimetria (ASYM), normal (NORM)
- Diagnòstics: benigne (B), maligne (M)
Atès que l'objectiu d'aquest projecte era determinar el millor llindar per a cada tipus de teixit de fons, no era necessària tota la informació. Tot i això, podeu ampliar el vostre projecte per incloure anàlisi de textures i provar el classificador amb les descripcions de massa conegudes.
Nota lateral: la base de dades de la qual vaig obtenir les imatges de mamografia diagnosticades va organitzar la informació sobre cada mamografia en un fitxer de text separat de les imatges. Em va resultar lleugerament difícil extreure les dades d'un fitxer de text i organitzar-les en formes de matriu, però el següent enllaç va ser molt útil per esbrinar tot això. També podeu ajustar el codi que he enganxat anteriorment per als vostres propòsits.
Format de fitxer de mamografia: mdb001 G CIRC B 535 425 197
mdb002 G CIRC B 522 280 69
Ajuda de TextScan: https://www.mathworks.com/help/matlab/ref/textsca… Base de dades de mamogrames:
Pas 2: processament d'imatges
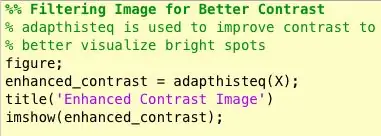
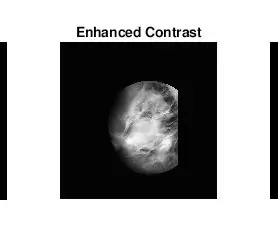
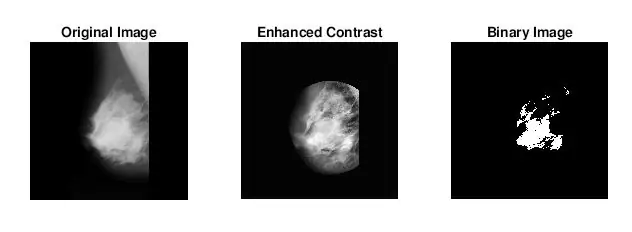
Bé, la segona cosa que va sorgir quan vaig esbrinar com identificar les masses va ser que, per a moltes mamografies anormals, no podia dir visualment on era l’anomalia ni la mida que tenia. Viouslybviament, com que no sóc un radiòleg experimentat, s’esperava. Tanmateix, la forma més senzilla de trobar anomalies (segons les meves llargues cerques a Google) era mirar concentracions de zones fosques i brillants. Vaig utilitzar principalment la funció adapthisteq per millorar el contrast de la imatge i després imbinar-la per convertir la imatge en una imatge binària per experimentar amb diferents nivells de llindar.
- adapthisteq: aquesta funció transforma els valors d’intensitat de les imatges en escala de grisos i rgb mitjançant l’equalització d’histogrames adaptatius de contrast limitat. En altres paraules, ajusta l'histograma dels valors d'intensitat a un tipus de distribució especificat. A continuació s’adjunta l’enllaç mathworks d’aquesta funció per a una lectura posterior.
- imbinar: crea una imatge binària a partir d’una imatge d’escala de grisos assignant tots els píxels superiors a una certa intensitat a 1s i els píxels per sota d’aquest valor a 0. He utilitzat aquesta funció per provar el llindar òptim per reduir el soroll del teixit de fons.
Pas 3: Codi de llindar
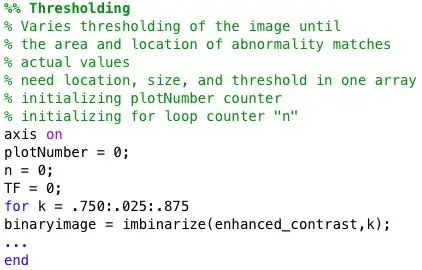
Un bucle for s'utilitza per binaritzar la mamografia amb llindars variables. Per donar una imatge més gran, el bucle for conté el codi del pas 3 al pas 7. Així, cada imatge binària serà analitzada per detectar anomalies. A més, aquest bucle for es troba encastat en un altre bucle for que importa una nova imatge de mamografia de la base de dades en cada iteració.
Pas 4: trobar anomalies per a cada imatge binària
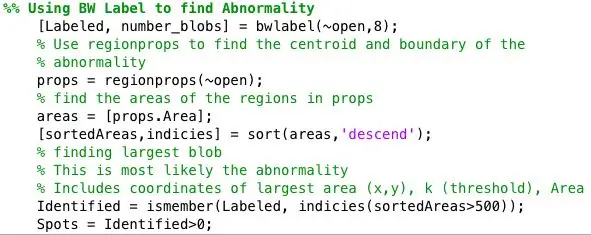
A més, he processat les imatges binàries mitjançant la funció strel juntament amb imopen per eliminar el soroll de fons. La imatge binària del pas anterior s’inverteix i es filtra utilitzant el veïnat definit per SE. Després vaig utilitzar bwlabel per etiquetar qualsevol àrea que tingués almenys 8 píxels connectats.
La funció de puntals de la regió es va utilitzar per trobar les propietats del centre i de l'àrea de cada punt identificat per bwlabel.
Llavors es van identificar tots els punts de més de 500 píxels mitjançant ismember. Els centroides dels punts identificats es van representar en una imatge que només mostrava els punts més grans que 500. Zona identificada = membre (Etiquetat, indicis (àrees ordenades> 500)); Spots = Identificat> 0;
Pas 5: representació de la ubicació i la mida de la massa diagnosticada per a la comparació visual
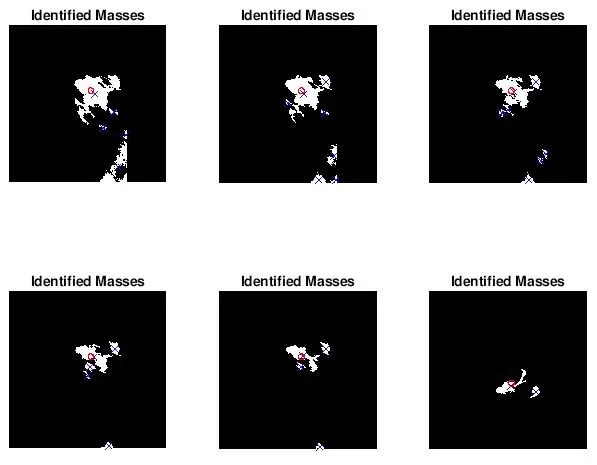
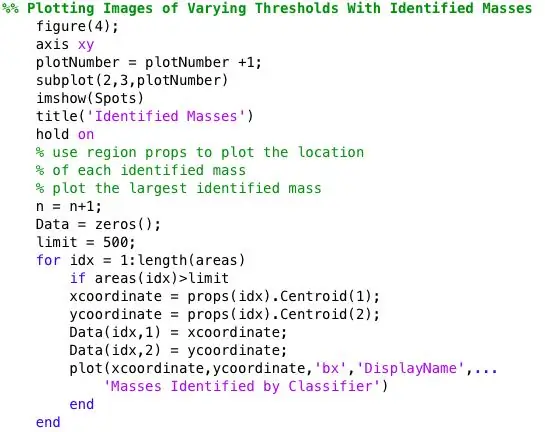

Volia veure si els punts trobats per bwlabel eren correctes. Ho vaig fer de dues maneres. Primer vaig analitzar la precisió del meu classificador fent una comparació visual. Simplement vaig representar la mida i la ubicació reals de l’anomalia (cercle vermell) i la ubicació determinada pel codi (x blau) a la imatge de la mamografia preprocessada. Les sis imatges anteriors mostren els efectes de l’augment del valor llindar en escala de grisos.
Pas 6: Implementació del segon mètode de comparació

La segona manera de provar el classificador i els valors llindars van ser determinant si les ubicacions trobades pel classificador es trobaven a una distància determinada de les coordenades d’anomalia diagnosticades. He desat els llindars dels quals almenys un dels punts identificats es trobava a 1,5 * r des de l’anomalia coneguda a un fitxer de text independent anomenat Dades de mamografia. El propòsit d’això era trobar el llindar mínim necessari perquè el meu classificador identifiqués l’anomalia.
Pas 7: analitzar les dades recollides
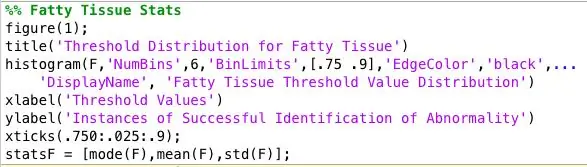
Vaig executar el programa amb totes les imatges mamogràfiques anormals i em va quedar un enorme fitxer de text de dades. Per tal de trobar el millor llindar per a cada tipus de teixit, vaig organitzar les dades per tipus de teixit i vaig representar un histograma dels valors llindars de cada tipus de teixit. Es va decidir el valor del llindar adequat sobre quin llindar proporcionava els resultats més precisos per a cada tipus de teixit. He desat aquestes dades per penjar-les al meu classificador.
Pas 8: Feu el vostre propi classificador

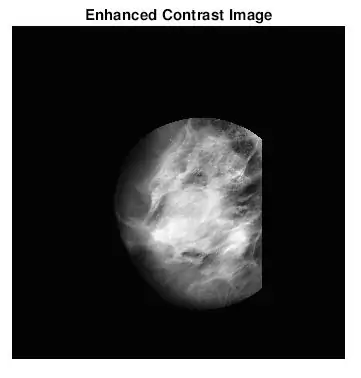
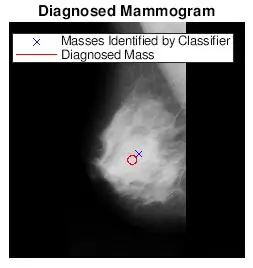
Després de trobar els valors llindars més adequats per a cada tipus de teixit, he editat el meu codi original perquè un usuari introdueixi el número de la imatge i el tipus de teixit per triar el llindar de la imatge de la mamografia. Després vaig representar la ubicació de la mamografia diagnosticada amb les ubicacions trobades a les imatges originals de la mamografia. Volia fer-ho més divertit, així que vaig programar una funció per retallar una regió circular que envolta el ROI. Es donaria instruccions a l’usuari per triar un punt central i diversos punts que millor englobin el ROI. He adjuntat aquí els dos fitxers matlab.
Pas 9: millores? Alguna idea?
Mentre escrivia aquest instructiu, començo a veure moltes millores que podia fer al classificador, com ara trobar maneres de distingir entre diferents tipus de masses identificades en funció de l’anàlisi de textures o millorar les meves proves de la secció de precisió del SandBoxProject. dossier. Com que es tractava d’un projecte amb una data límit, vaig haver d’aturar-me en algun lloc, però espero poder utilitzar les habilitats de processament d’imatges que he après en altres aplicacions. A més, he adjuntat el fitxer que s’utilitzava per processar per lots totes les imatges mamogràfiques anormals.
Recomanat:
Intensitat del senyal WiFi ESP32 TTGO: 8 passos (amb imatges)

Intensitat del senyal WiFi ESP32 TTGO: en aquest tutorial aprendrem a mostrar la intensitat del senyal de la xarxa WiFi mitjançant la placa ESP32 TTGO. Mireu el vídeo
Millor manera de visualitzar / documentar fitxers en instruccions: 4 passos
Millor manera de visualitzar / documentar fitxers a instructables: la gent sovint hi inclou fitxers per a projectes mitjançant càrregues d’Instructables. Sovint es tracta de fitxers font de programari que poden ser fins i tot el cor de l’instructible. Però Instructables no proporciona al lector una manera fàcil de llegir i revisar el codi. (
Temporitzador d’entrenament per intervals d’alta intensitat (HIIT): 3 passos

Temporitzador d’entrenament per intervals d’alta intensitat (HIIT): on visc, els mesos freds semblen durar per sempre, de manera que he de trobar alguna manera d’exercitar-me que em mantingui dins de casa. Em podia permetre el luxe d’anar a un gimnàs, però em costa massa temps, hauria de mostrar el meu vell cos en públic i no seria capaç de veure
Mesurador d'intensitat lluminosa sense programació .: 7 passos (amb imatges)

Mesurador de la intensitat de la llum sense programació. Aquesta instrucció es tracta de fer un mesurador bàsic de la intensitat de la llum sense fer servir Arduino ni cap altre microcontrolador ni programació. El mesurador d'intensitat de llum mostra diferents nivells d'intensitat de llum amb diferents colors de LED. El LED vermell
Circuit de llum de vela OLED amb fotoresistència per al control de la intensitat (TfCD): 4 passos (amb imatges)

Circuit de llum d’espelmes OLED amb fotoresistència per al control d’intensitat (TfCD): en aquest instructiu us mostrem com fer un circuit que mostri el LED (O) que parpelleja com una espelma i reaccioni a la intensitat de l’entorn. Amb una intensitat de llum més baixa es necessita una sortida de llum més baixa de les fonts de llum. Amb aquesta aplicació
