Taula de continguts:
- Autora John Day day@howwhatproduce.com.
- Public 2024-01-30 08:14.
- Última modificació 2025-01-23 14:38.
Per: Madhumita Kannan, Henry Nguyen, Ashley Urrutia Avila, Mei Jin
Aquest codi MATLAB és un programa per detectar la mida, la forma i la ubicació exactes d’un tumor que es troba a la ressonància magnètica del pacient. Aquest programa està dissenyat per treballar originalment amb la detecció de tumors en exploracions de ressonància magnètica cerebral, però també es pot utilitzar per al diagnòstic del càncer en altres exploracions d’òrgans.
Les instruccions següents descriuen primer els mètodes d’anàlisi d’imatges mitjançant el filtratge i la neteja de l’exploració de ressonància magnètica, mitjançant la binarització, el filtratge mitjà i les finestres corredisses. A continuació, s’informarà sobre com aïllar el tumor mitjançant una màscara el·líptica pre-generada i es filtrarà més per esbossar el perímetre de la forma del tumor.
Un cop detectat el tumor, les instruccions descriuran com incorporar aquest programa a una interfície gràfica d'usuari (GUI). Al llarg d’aquestes instruccions, s’adjuntaran el codi i els fitxers adequats per ajudar a explicar com funciona aquesta anàlisi de ressonància magnètica.
Algunes coses que heu de saber, descarregar i tenir a punt abans de continuar amb aquesta instrucció: 1. Assegureu-vos de descarregar la versió més recent de MATLAB. Podeu instal·lar R2018b aquí:
2. Per executar aquest programa, heu de tenir accés als fitxers d’exploració cerebral de ressonància magnètica. Tot i que sempre es poden trobar algunes a partir d’imatges de Google, es poden realitzar anàlisis exhaustives i precises a partir d’imatges adequades de diverses capes d’exploracions cerebrals per a cada pacient. Podeu accedir als fitxers de 20 pacients diferents amb glioblastoma abans i després del tractament des d’aquesta base de dades:
3. L’enfocament d’aquest programa i els diversos mètodes que guien aquest projecte es descriuen en aquest treball de recerca:
Pas 1: Inicieu la interfície gràfica d'usuari (GUI)
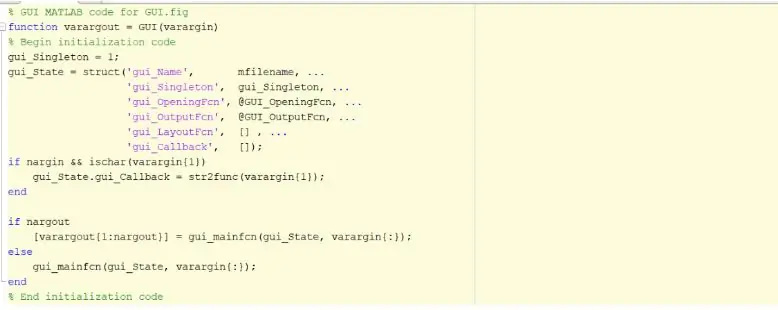
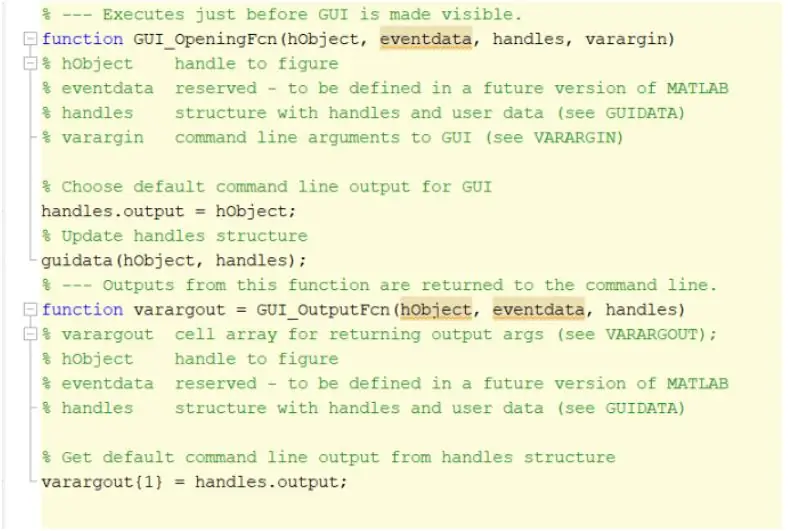
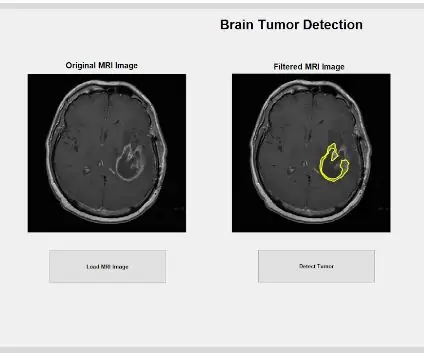
El primer pas seria crear i iniciar la interfície gràfica d’usuari, GUI. Això es podria fer escrivint guia a la finestra d'ordres, prement Intro i creant una nova interfície gràfica d'usuari. Un cop completat aquest pas, podeu començar a crear funcions com ara eixos, text estàtic, text d'edició i botons que es mostraran un cop s'executi el programa i l'usuari pugui interactuar. Aquestes funcions es poden editar i manipular mitjançant l’inspector de propietats, però la característica més important que s’ha d’alterar en crear aquestes funcions és el nom de l’etiqueta. És important canviar el nom de l'etiqueta de cada funció que s'implementa perquè ens permetrà crear una funció de devolució de trucada que es distingeixi. Quan estigueu satisfet amb el disseny de la vostra interfície gràfica d’usuari, podeu passar a carregar els fitxers DICOM que es mostraran a la interfície gràfica d’usuari.
Pas 2: carregar i llegir imatges de ressonància magnètica a MATLAB
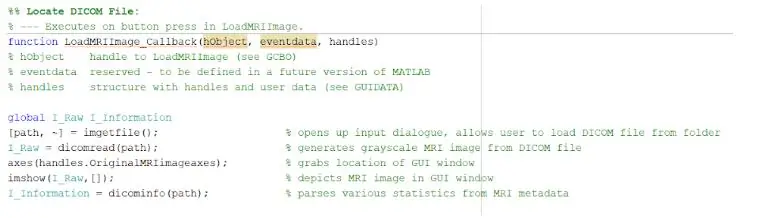
Per carregar els fitxers DICOM, haureu d'iniciar correctament la funció de devolució de trucada que s'executaria en prémer el botó "Carrega imatge MRI". Un cop s'hagi completat, heu de crear una variable global que mostri la imatge als eixos de controladors on vulgueu que es mostri la imatge de ressonància magnètica original. Les imatges d’exploració de ressonància magnètica descarregades de la base de dades són fitxers amb format DICOM que cal carregar al directori MATLAB. Localitzeu el fitxer amb imgetfile per tal de carregar-los al programa. Les imatges es llegeixen mitjançant la funció MATLAB integrada "dicomread" i la primera imatge en brut de cada fitxer s'incrusta als eixos de la GUI esquerra mitjançant imshow.
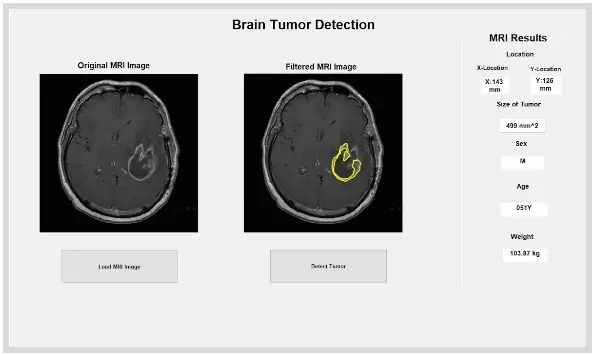
La funció MATLAB incorporada "dicominfo" també és extremadament útil per tractar tota la informació de cada fitxer dicom de ressonància magnètica. Hem utilitzat aquesta funció per extreure tota la informació descriptiva dels pacients, com ara el seu sexe, edat, pes i alçada. Aquesta funció també us proporciona l'ordre de pila que és útil per a la implementació del programa a la interfície gràfica d'usuari. Hem creat variables per a cadascuna de la informació descriptiva dels pacients que s’utilitzaran per a la GUI quan es prem el botó de detecció.
Pas 3: filtratge d'imatges

Un cop s'ha carregat i llegit el fitxer DICOM de la imatge en brut, la imatge ha de convertir-se en escala de grisos a una forma binaritzada que consisteix només en píxels en blanc i negre. Hem utilitzat la funció "imbinar" per crear una imatge binària a partir de la imatge en brut aspectes de control del llindar adaptatiu amb un valor de sensibilitat de 0,59. El factor de sensibilitat del llindar per defecte, 0,5, era baix i no es podia detectar les taques i els punts més brillants de la imatge, de manera que l’hem augmentat a 0,59.
La imatge binaritzada es processa a través d’un filtre mitjà mitjançant la funció ‘medfilt2’ perquè la imatge binaritzada és bidimensional. Establim cada píxel de sortida perquè contingui el valor mitjà al barri de 5 x 5 al voltant del píxel corresponent de la imatge binaritzada d’entrada. Això redueix el soroll i preserva les vores en un quadrat de 5 x 5 al voltant de cada píxel. A continuació, apliquem una finestra lliscant amb ‘strel’, per crear un element d’estructuració pla en forma de disc amb un radi de veïnatge de 2 per identificar cada píxel d’origen central a cada barri del disc. Hem utilitzat un element d’estructuració de disc perquè estem analitzant cada punt circular i els píxels dins de cada lloc, de manera que un element de forma de disc és més útil.
Un cop filtrada la imatge, es pot netejar mitjançant la funció "imclose" per eliminar les taques negres que hi ha entre els píxels blancs filtrats de la imatge i es tanquen tots els buits al seu voltant. La imatge completament processada es pot dibuixar a la segona subtrama de la figura assignada prèviament, permetent una comparació entre la imatge en brut i filtrada.
Pas 4: Aïllament del tumor mitjançant la màscara el·líptica


Els punts brillants del tumor es poden aïllar de la imatge filtrada principal mitjançant una màscara el·líptica pre-generada. Per crear aquesta màscara, heu de conèixer la mida de la imatge d’exploració de ressonància magnètica en brut i original i, mitjançant la longitud de la fila i la columna, com a coordenades x i y, respectivament, assigneu les coordenades centrals per a l’el·líptica. Establim l'eix y com a eix principal amb un radi de 50 unitats del centre i l'eix menor amb un radi de 40 unitats des del centre.
Hem utilitzat la funció MATLAB 'meshgrid' per generar un pla cartesià amb coordenades de quadrícula bidimensionals basades en les coordenades contingudes en vectors d'1 a la longitud de l'eix x i d'1 a la longitud de l'eix y de la imatge. Col és una matriu en què cada fila és una còpia de l’eix x i Row és una matriu en què cada columna és una còpia de l’eix y. La quadrícula cartesiana representada per les coordenades Col i Row té files de longitud (1: Y_Size) i columnes de longitud (1: X_Size). Utilitzeu els índexs de Col i Row generats per la quadrícula cartesiana per determinar l’equació de l’el·lipse en funció del radi i les coordenades centrals predeterminades. El contorn el·líptic ara es pot omplir amb els píxels blancs que es troben a les taques tumorals.
Mitjançant la màscara el·líptica pre-generada podem retallar el tumor específic que voleu analitzar a partir de la imatge filtrada. La màscara el·líptica detecta quins punts encaixen lògicament dins del contorn de l’el·lipse i l’accepta com un punt de la imatge filtrada per ser acceptable com a tumor. A continuació, la funció ‘bwareafilt’ filtra de la imatge tots els altres objectes fora d’aquest tumor detectat. Hem utilitzat una finestra específica de 500 per 4000 empíricament en funció de les dimensions de totes les imatges. A continuació, vam aplicar una altra finestra corredissa amb ‘strel’ com a element estructurant en forma de disc pla d’un radi de veïnatge més gran de 6, per tancar els buits entre cada píxel blanc central dins del tumor detectat. La taca tumoral detectada es neteja encara més amb ‘imclose’ per eliminar encara més els píxels negres i omplir tots els forats amb ‘imfill’. Aquest tumor processat es pot mostrar a la tercera subtrama de la trama preasignada per proporcionar una comparació entre el tumor aïllat i les imatges originals i filtrades de la ressonància magnètica.
Pas 5: Esquema del tumor
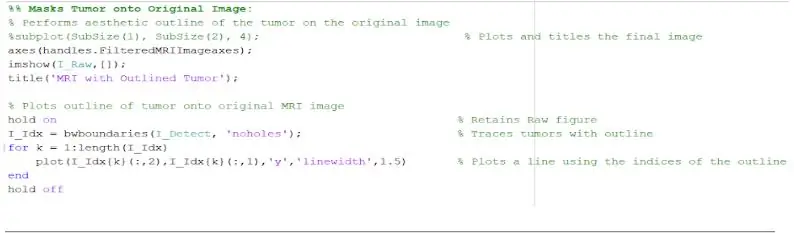
Ara que el tumor està aïllat amb la màscara, es pot esbossar i mostrar a la imatge original per mostrar la seva ubicació exacta. Per fer-ho, hem utilitzat la funció "bwboundaries" per rastrejar el tumor detectat anteriorment amb un esquema. Vam especificar l'esquema per no incloure els forats dins de l'objecte tumoral tal com es perfila. Això es pot representar a la imatge original, en brut, mitjançant un bucle "for" que representa el contorn al voltant del tumor mitjançant els índexs de la línia amb una amplada de línia de 1,5 píxels. Aquest esbós es representa a la imatge bruta, mostrant la mida i la ubicació exactes del tumor, en relació amb la ressonància magnètica original.
Pas 6: analitzar les propietats físiques dels tumors
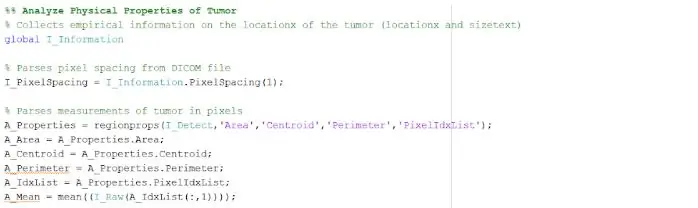
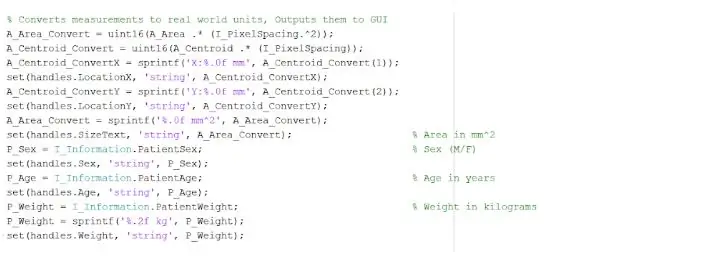
El punt aïllat i esbossat ens pot proporcionar informació útil sobre la mida, l’àrea i la ubicació del tumor. Hem utilitzat la funció ‘regionprops’ per detectar les propietats del tumor que pertanyen a l’àrea, el perímetre, els centroides i el valor de l’índex de píxels. Aquest valor de l’índex de píxels ens proporciona les unitats del món real de cada píxel de cada imatge, exclusives de cada exploració. Aquestes propietats es poden convertir en unitats reals de mil·límetres. La informació empírica que ens proporciona el programa és única per a cada ressonància magnètica i és extremadament útil per determinar la mida, la ubicació i el tipus de tumor que els usuaris poden analitzar i incorporar a la interfície gràfica d’usuari.
Recomanat:
Com fer un timbre sense tocar, detecció de temperatura corporal, GY-906, 433 MHz mitjançant Arduino: 3 passos
Com fer un timbre sense tocar, detecció de temperatura corporal, GY-906, 433 MHz utilitzant Arduino: Avui farem un timbre sense tocar, que detectarà la temperatura corporal. En la situació actual, és molt important saber si la temperatura corporal d'algú és superior a la normal, quan algú està tocant. Aquest projecte mostrarà llum vermella si es detecta
Càmera IP amb detecció de cares mitjançant la placa ESP32-CAM: 5 passos

Càmera IP amb detecció de cares mitjançant la placa ESP32-CAM: aquesta publicació és diferent en comparació amb les altres i fem una ullada a la molt interessant placa ESP32-CAM que és sorprenentment barata (menys de 9 dòlars) i fàcil d’utilitzar. Creem una càmera IP senzilla que es pot utilitzar per transmetre un canal de vídeo en directe mitjançant el 2
Detecció senzilla de colors mitjançant OpenCV: 6 passos
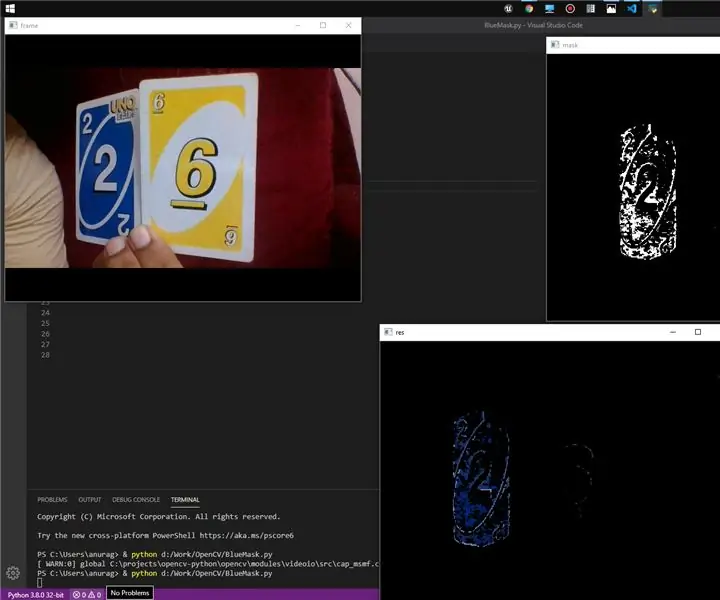
Detecció senzilla de colors mitjançant OpenCV: Hola! Avui mostraré un mètode senzill per detectar un color d’un vídeo en directe mitjançant OpenCV i python. Bàsicament, només provaré que el color requerit estigui present o no en el marc de fons i amb els mòduls OpenCV emmascararé aquesta regió i
Detecció de color a Python mitjançant OpenCV: 8 passos
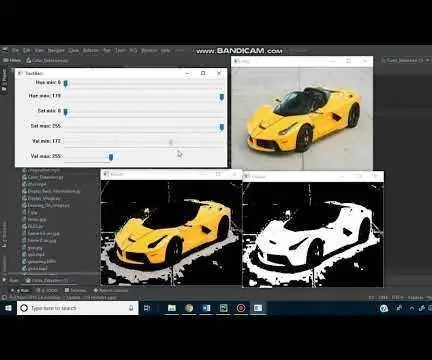
Detecció de colors a Python mitjançant OpenCV: Hola! Aquesta instrucció s'utilitza per guiar sobre com extreure un color específic d'una imatge en python mitjançant la biblioteca openCV. Si no coneixeu aquesta tècnica, no us preocupeu, al final d'aquesta guia podreu programar el vostre propi color
Una caixa de ressonància de butxaca: 6 passos

A Pocket Soundbox: aquest dispositiu no només s'adapta a una butxaca, sinó que també produeix diversos tons musicals similars als d'una gaita (al meu entendre) mitjançant diverses combinacions de sis botons. Viouslybviament, només és un aparell per divertir els nens; tanmateix, és principi
