Taula de continguts:
- Autora John Day day@howwhatproduce.com.
- Public 2024-01-30 08:11.
- Última modificació 2025-01-23 14:37.
Per: Phuc Lam, Paul Yeung, Eric Reyes
Sabent que els errors en la segmentació dels pulmons produiran informació falsa sobre la identificació d’una zona de malaltia i poden afectar directament el procés de diagnòstic. Les tècniques modernes d’ajuda per ordinador no van aconseguir resultats precisos quan les malalties pulmonars tenen formes difícils. Aquestes formes anormals poden ser causades per efusions pleurals, consolidacions, etc. Aplicant la tècnica de segmentació pulmonar, en què els límits del pulmó s’aïllen del teixit toràcic que l’envolta, la nostra aplicació pot identificar els límits amb els llindars d’entrada de l’usuari per obtenir vistes totalment personalitzables. de les formes dels pulmons, L’objectiu d’aquest projecte MatLab és crear una aplicació interactiva de segmentació pulmonar fàcil d’utilitzar per detectar les condicions patològiques de les imatges de raigs X dels pulmons. El nostre objectiu és crear una manera més eficaç d’il·lustrar i identificar pulmons anormals per donar als metges i als radiòlegs una manera més fiable de diagnosticar malalties pulmonars. Mitjançant l’eina de disseny d’aplicacions de MatLab, el programa està dissenyat per treballar específicament amb exploracions de radiografia de tòrax i tomografia computada (TC), però també es prova per funcionar amb exploracions de ressonància magnètica.
Les instruccions següents contenen la nostra tècnica de filtrat de soroll (filtre Wiener de pas baix), així com el llindar de la imatge (mitjançant l’histograma d’intensitat de la imatge en escala de grisos) i mitjançant un gradient morfològic (la diferència entre la dilatació i l’erosió d’una imatge) per identificar una regió d’interès. A continuació, la instrucció explicarà com integrem tots els elements a la interfície gràfica d'usuari (GUI).
Nota:
1). Aquest projecte s’inspira en un article de recerca: "Segmentació i anàlisi d’imatges de pulmons anormals a la TC: enfocaments actuals, desafiaments i tendències futures". Que es pot trobar aquí
2). Estem utilitzant imatges de raigs X de NIH: Clinical Center. L’enllaç es pot trobar aquí
3). Podeu trobar ajuda per al dissenyador d'aplicacions aquí
4). Abans d’executar el codi: heu de canviar la ruta Dir (a la línia 34) al directori de fitxers i el tipus d’imatge (línia 35) (estem analitzant *.png).
Pas 1: pas 1: càrrega de la imatge
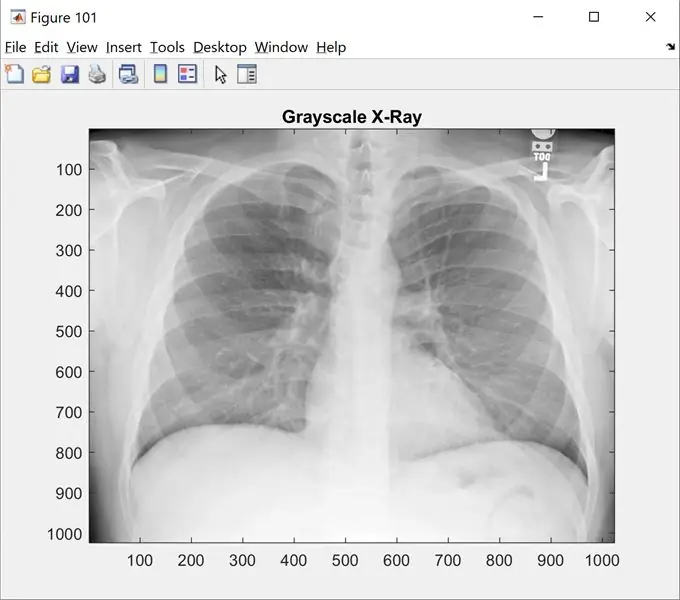
Aquest pas us mostrarà la imatge original a escala de grisos. Canvieu el "nom_de_imatge.png" pel nom de la vostra imatge
clar; clc; tancar-ho tot;
%% Carregant imatges
raw_x_ray = 'nom_de_imatge.png';
I = imread (raw_x_ray);
figura (101);
imshow (I);
mapa de colors (gris);
title ('Radiografia en escala de grisos');
Pas 2: Pas 2: filtratge de soroll i histograma
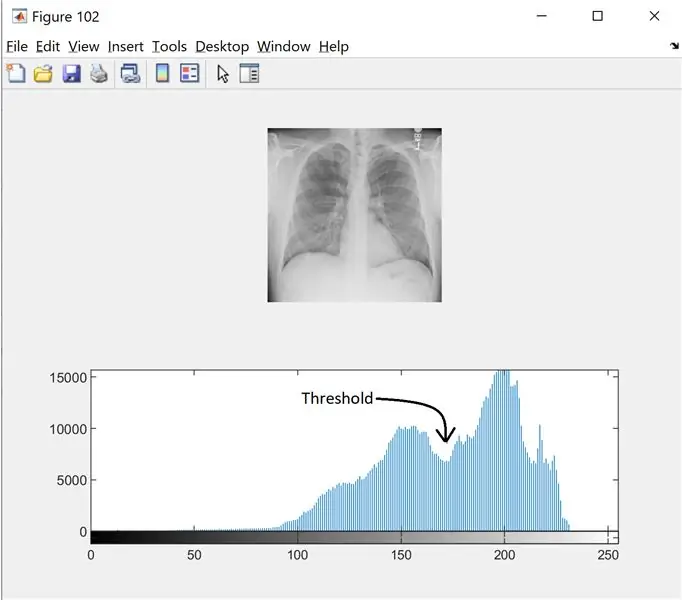
Per tal de trobar el llindar de la imatge a escala de grisos, mirem l'histograma per veure si hi ha modes diferents. Llegiu-ne més aquí
I = wiener2 (I, [5 5]);
figura (102);
subtrama (2, 1, 1);
imshow (I);
subtrama (2, 1, 2);
imhist (I, 256);
Pas 3: Pas 3: Configuració de llindars
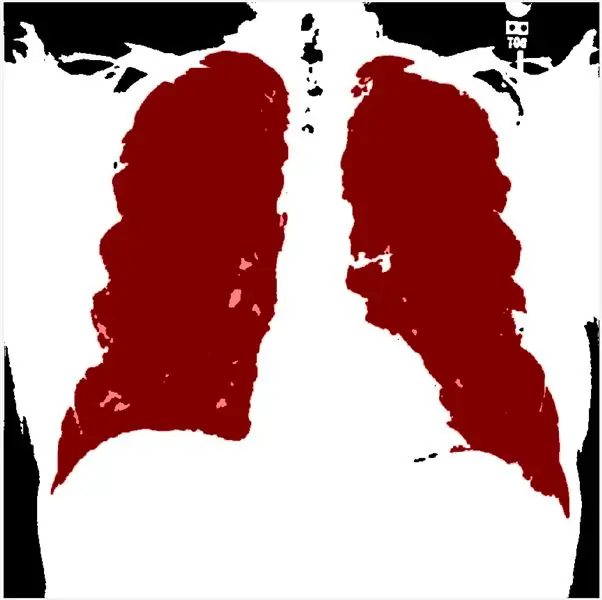
Aquest pas us permet establir el llindar segons l'histograma. morphologicalGradient ressaltarà la regió d’interès en vermell i la funció visboundaries recobreix la imatge esbossada i filtrada del pulmó en vermell.
Mitjançant l’ús de regionalprops, podem aplicar les matrius de solidesa i ordenar-les de manera descendent. A continuació, binaritzo la imatge de sclae gris i aplico el mètode de gradient morfològic i mLoren Shurasking per ressaltar la regió d’interès (ROI). El següent pas és invertir la imatge perquè el ROI del pulmó sigui blanc en el fons negre. Utilitzo la funció showMaskAsOverlay per mostrar 2 màscares. Nota: el codi està inspirat en Loren Shure, enllaç.
Lasly, creo un esquema vermell mitjançant l'ús de bwbwboundaries i emmascarant la imatge del filtre i els límits.
a_thresh = I> = 172; % ha establert aquest llindar
[labelImage, numberOfBlobs] = bwlabel (a_thresh);
props = regionprops (a_thresh, 'all');
sortedSolidity = ordenar ([props. Solidity], 'descendir');
SB = ordenat Solidesa (1);
si SB == 1% SB només accepta solidesa == 1 filtra els ossos
binaryImage = imbinar (I); figura (103);
imshow (binaryImage); mapa de colors (gris);
SE = strel ("quadrat", 3);
morphologicalGradient = imsubtract (imdilate (binaryImage, SE), imerode (binaryImage, SE));
màscara = imbinar (morfologicalGradient, 0,03);
SE = strel ('quadrat', 2);
màscara = imclose (màscara, SE);
màscara = omplir (màscara, 'forats');
màscara = bwareafilt (màscara, 2); % nombre de control de mostra de l'àrea
notMask = ~ màscara;
màscara = màscara | bwpropfilt (notMask, 'Area', [-Inf, 5000 - eps (5000)]);
showMaskAsOverlay (0,5, màscara, 'r'); % heu de descarregar l'aplicació / funció showMaskAsOverlay
BW2 = imfill (binaryImage, 'forats');
nova_imatge = BW2;
nova_imatge (~ màscara) = 0; % de fons i forats invertits
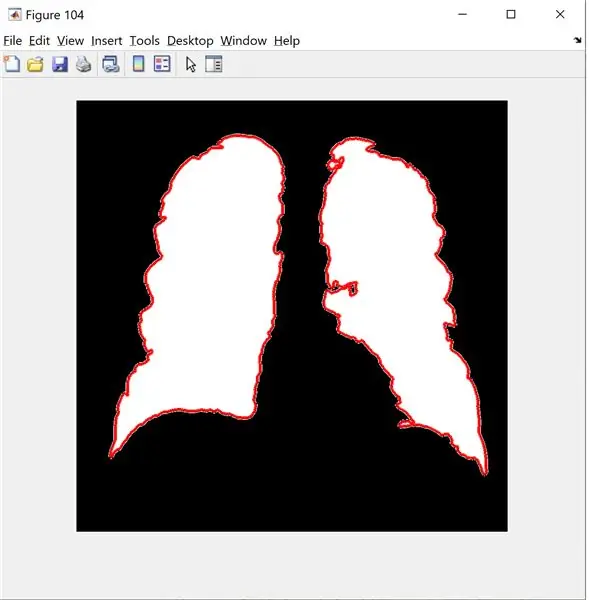
B = fronteres bw (nova_imatge); % només pot acceptar 2 dimensions
figura (104);
imshow (nova_imatge);
espera
límits viscuts (B);
final
Pas 4: creació de la GUI
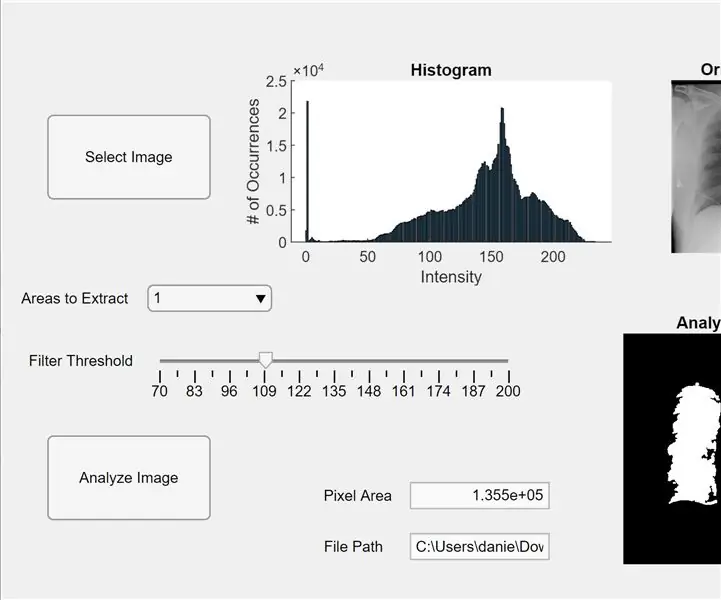
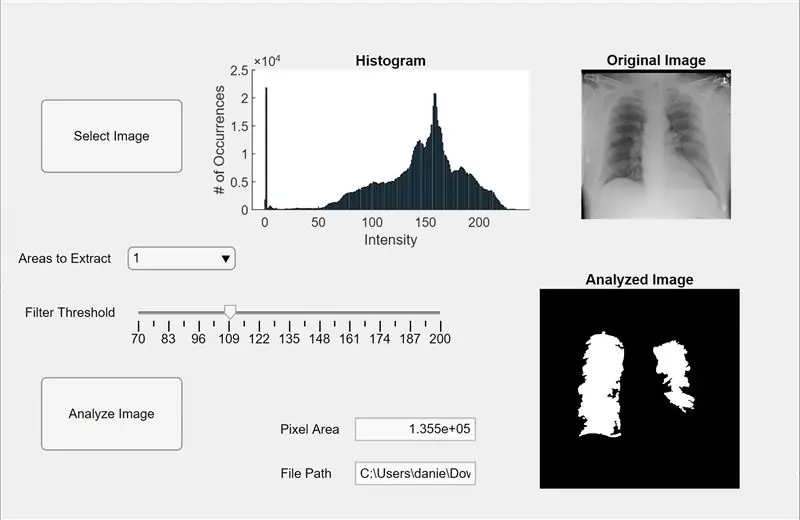
Ara, integrem el codi anterior en una aplicació MATLAB. Obriu el Dissenyador d'aplicacions a MATLAB (Nou> Aplicació). En primer lloc, dissenyem la interfície fent clic i mantenint premut i arrossegant tres eixos a l’espai de treball central. A continuació, fem clic-mantingueu-arrossegueu dos botons, un camp d'edició (text), un camp d'edició (numèric), un control lliscant i un menú desplegable. Dos eixos mostraran cadascun la vista prèvia i analitzaran la imatge, i els tercers eixos mostraran un histograma de píxels per a la imatge "seleccionada" de vista prèvia. El quadre d'edició (text) mostrarà la ruta del fitxer de la imatge seleccionada i el camp d'edició (numèric) mostrarà l'àrea de píxels detectada dels pulmons.
Ara canvieu de la visualització de disseny a la de codi a App Designer. Introduïu al codi el codi de les propietats fent clic al botó vermell "Propietats" amb el signe més al seu costat. Inicialitzeu les propietats I, el llindar i regionsToExtract tal com es mostra al codi que es proporciona a continuació. A continuació, feu clic amb el botó dret a un botó a la part superior dreta de l'espai de treball (el navegador de components) i aneu des de Devolucions de trucades> Ves a … devolució de trucada. Afegiu el codi per a la "funció SelectImageButtonPush (aplicació, esdeveniment)". Aquest codi us permet seleccionar una imatge per analitzar des del vostre ordinador mitjançant uigetfile. Després de seleccionar una imatge, apareixerà una imatge de previsualització sota els eixos acompanyada d’un histograma. A continuació, feu clic amb el botó dret a l’altre botó i repetiu el mateix procediment per crear una funció de devolució de trucada.
Afegiu el codi que hi ha a sota de la "funció AnalyzeImageButtonPush (aplicació, esdeveniment)". Aquest codi realitzarà el recompte de píxels i la detecció de blob a la imatge de previsualització amb el botó d'anàlisi de la imatge (el que hagueu fet clic amb el botó dret del ratolí per obtenir aquest codi). Després de programar els botons, ara programarem el control lliscant i el menú desplegable. Feu clic amb el botó dret del ratolí sobre el control lliscant, creeu una funció de devolució de trucada i afegiu-hi el codi a sota de la funció "FilterThresholdSliderValueChanged (app, event)" fins al final. Això permet al control lliscant ajustar el llindar d’intensitat de grisos.
Creeu una funció de devolució de trucada per al menú desplegable i afegiu-hi el codi que apareix a sota de la "funció AreastoExtractDropDownValueChanged (aplicació, esdeveniment)" per permetre al menú desplegable modificar el nombre de blobs que es mostren als eixos de la imatge analitzada. Ara, feu clic a cada entitat al navegador de components i canvieu les seves propietats al vostre gust, com ara canviar els noms de les entitats, eliminar eixos i canviar l'escala. Arrossegueu i deixeu anar les entitats del navegador de components a la vista de disseny fins a un disseny funcional i fàcil d'entendre. Ara teniu una aplicació a MATLAB que pot analitzar imatges de pulmons per a l'àrea de píxels.
propietats (Accés = privat) I = ; % fitxer d'imatge
llindar = 257; % llindar per binaritzar la intensitat del gris
regionsToExtract = 2;
final
funció SelectImageButtonPush (aplicació, esdeveniment)
clc; Dir = 'C: / Users / danie / Downloads / images_004 / images'; % defineix el fitxer invariable "prefix"
[imageExt, path] = uigetfile ('*. png'); % agafa la part variable del nom de la imatge
imageName = [Dir fitxersep imageExt]; % concatenar cordons invariables i variables
app. I = imread (imageName); % llegeix la imatge
imshow (app. I, 'pare', app. UIAxes); % mostra la imatge
app. FilePathEditField. Value = ruta; Percentatge de visualització del fitxer% d'on prové la imatge original
final
funció AnalyzeImageButtonPush (aplicació, esdeveniment)
originalImage = app. I;
Imatge original = wiener2 (app. I, [5 5]); % filtre d'eliminació de punts
histograma (app. AxesHistogram, app. I, 256); % mostra l'histograma de la imatge
a_thresh = originalImage> = app.threshold; % ha establert aquest llindar
labelImage = bwlabel (a_thresh);
props = regionprops (a_thresh, 'all');
sortedSolidity = ordenar ([props. Solidity], 'descendir');
SB = ordenat Solidesa (1);
si SB == 1% SB només accepta solidesa == 1 filtra els ossos
SE = strel ("quadrat", 3);
morphologicalGradient = imsubtract (imdilate (labelImage, SE), imerode (labelImage, SE));
màscara = imbinar (morfologicalGradient, 0,03);
SE = strel ('quadrat', 2);
màscara = imclose (màscara, SE);
màscara = omplir (màscara, 'forats');
màscara = bwareafilt (màscara, app.regionsToExtract);
% nombre de control de mostra de l'àrea
notMask = ~ màscara;
màscara = màscara | bwpropfilt (notMask, 'Area', [-Inf, 5000 - eps (5000)]);
BW2 = emplenament (labelImage, 'forats');
nova_imatge = BW2;
nova_imatge (~ màscara) = 0;
B = fronteres bw (nova_imatge); % només pot acceptar imshow de 2 dimensions (new_image, 'parent', app. UIAxes2);
hold (app. UIAxes2, 'on');
límits viscuts (B);
set (gca, 'YDir', 'revers');
lungArea = bwarea (nova_imatge);
app. PixelAreaEditField. Value = LungArea;
final
final
funció FilterThresholdSliderValueChanged (aplicació, esdeveniment)
app.threshold = app. FilterThresholdSlider. Value;
final
funció AreastoExtractDropDownValueChanged (aplicació, esdeveniment) stringNumber = app. AreastoExtractDropDown. Value;
app.regionsToExtract = str2double (stringNumber);
final
final
Recomanat:
MATLAB Easy Face Detection: 4 passos

MATLAB Easy Face Detection: l’objectiu principal d’aquest instructiu és mostrar el fàcil que serà el processament d’imatges. Amb l’ajut de MATLAB, la detecció i el seguiment de la cara han estat un camp de recerca important i actiu, per això vaig a explicar com es pot fer amb enginy
Com codificar i publicar Matlab 2016b a Word (Guia per a principiants): 4 passos

Com codificar i publicar Matlab 2016b a Word (Guia per a principiants): Matlab és un programa de llenguatge d’alt rendiment que s’utilitza per calcular resultats tècnics. Té la capacitat d’integrar visuals, càlculs i programació d’una manera fàcil d’utilitzar. Amb aquest programa, l'usuari pot publicar problemes i solucions
Control de Raspberry Pi mitjançant Matlab: 5 passos

Control de Raspberry Pi mitjançant Matlab: Ei, aquest tutorial tracta de controlar el vostre raspberry pi sense cap mitjançant matlab. És possible que hagueu d’instal·lar matlab a la versió més recent per donar suport a la nova placa de raspberry pi
Controlador robòtic ROS basat en Matlab: 9 passos
Controlador robotitzat ROS basat en Matlab: des de petit, sempre he somiat amb ser Iron Man i encara ho faig. Iron Man és un d'aquests personatges realment possibles i, en poques paraules, aspiro a ser Iron Man algun dia, fins i tot si la gent es riu de mi o diu que és impossible
Millores de programació de trens a MATLAB: 6 passos

Millores de programació de trens a MATLAB: Objectiu: l’objectiu d’aquest sistema programat és mirar Arduino a petita escala i aplicar la codificació a una escala més gran per millorar potencialment certes característiques de seguretat dels sistemes ferroviaris Amtrak. Per fer-ho, hem afegit un sen
