Taula de continguts:
- Pas 1: fer servir Brain Box
- Pas 2: Introducció: analitzar seccions transversals
- Pas 3: Configuració d'una funció: LevelCurveTracings.m
- Pas 4: Desenvolupament de l'algorisme Bound: Cerca de Bounds
- Pas 5: Desenvolupament de l'algorisme Bound: generació de matriu de Bound exterior
- Pas 6: Desenvolupament de l'algorisme Bound: Treballar amb el punt central
- Pas 7: Desenvolupament de l'algorisme Bound: quan un Centroid no està centrat
- Pas 8: Desenvolupament de l'algorisme Bound: interferència de forats
- Pas 9: Desenvolupament de l'algorisme Bound: localització de forats, finalització de límits cerebrals i forats
- Pas 10: registre de dades: funció PatientFiles.m
- Pas 11: registre de dades als fitxers
- Pas 12: registre de dades: visualització d'una parcel·la de volum cerebral al llarg del temps
- Pas 13: Tancament de les llacunes a les subtrames: Subplotclose.m
- Pas 14: el codi principal: esborrar-ho tot i sol·licitar entrades
- Pas 15: el codi principal: processament per lots de les imatges
- Pas 16: el codi principal: encoixinat
- Pas 17: el codi principal: determinar límits
- Pas 18: el codi principal: determinar la dimensió Z adequada
- Pas 19: el codi principal: determinar les coordenades X i Y
- Pas 20: el codi principal: traçar una estructura tridimensional, trobar volum i registrar dades
- Autora John Day day@howwhatproduce.com.
- Public 2024-01-31 10:17.
- Última modificació 2025-01-23 14:38.
L’avanç cap a la frontera d’una vida humana més llarga ha provocat l’aparició de malalties que les civilitzacions no han vist abans que la nostra. Entre aquests, l’Alzheimer va afectar aproximadament 5,3 milions d’americans ancians vius el 2017, o aproximadament 1 de cada deu nord-americans ancians (https://www.alz.org/facts/) i innombrables altres persones amb demència. Per ajudar a la lluita per entendre què està afectant els nostres ancians, aquest codi equiparà els futurs investigadors i curiosos desitjosos amb la capacitat de rastrejar el volum del cervell al llarg del temps.
Pas 1: fer servir Brain Box
Per utilitzar la caixa del cervell, només cal el següent:
- Exploracions per ressonància magnètica d’un cervell i el nom i el format d’aquests fitxers (tots haurien de tenir aproximadament les mateixes dimensions)
- Durada d'una exploració
- Distància entre cada capa (ressonància magnètica)
- Nom del pacient (quan introduïu, no inclogueu espais i poseu majúscules en nom i cognom, com ara: nom i cognom)
I a partir d’això, es té la capacitat de fer un seguiment de les tendències dels individus en el volum del cervell al llarg del temps. Per tant, aquest programari pot fer un seguiment de les xifres de les tendències de l’Alzheimer. La longitud que vam fer servir a la prova va ser de 180 mm per a la longitud d’un escanejat i de 5 mm per a la distància entre exploracions de ressonància magnètica, segons les xifres mitjanes.
No obstant això, l'aplicació de la caixa cerebral no ha de limitar-se a aquesta tasca. Si les seccions transversals d’un sòlid donat són fotografies, com un tumor per se, les tendències en els canvis de volum d’aquests també es poden fer un seguiment al programari.
Pas 2: Introducció: analitzar seccions transversals
En estructures tridimensionals, els plans bidimensionals que consten d’aquestes s’anomenarien seccions transversals. Imagineu-vos que una pila de papers constitueix un prisma rectangular i que cada tros de paper seria una secció transversal del paper. En imaginar el cervell, apliquem el mateix curs de pensament. La ressonància magnètica (ressonància magnètica) (veure informació sobre la ressonància magnètica) capta les seccions transversals del cervell i, a partir de l’ús dels límits definits en cada “capa” del cervell proporcionada, podem construir una estructura per modelar i trobar el volum del cervell. En primer lloc, hem de construir una funció per proporcionar informació sobre aquests límits.
Pas 3: Configuració d'una funció: LevelCurveTracings.m
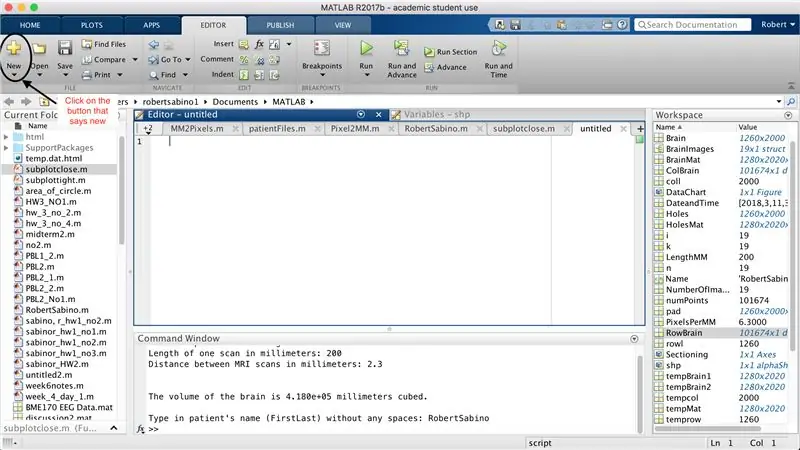
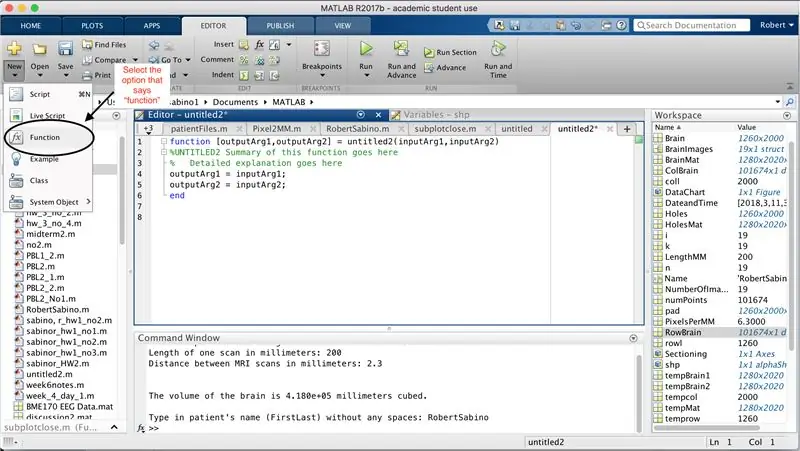
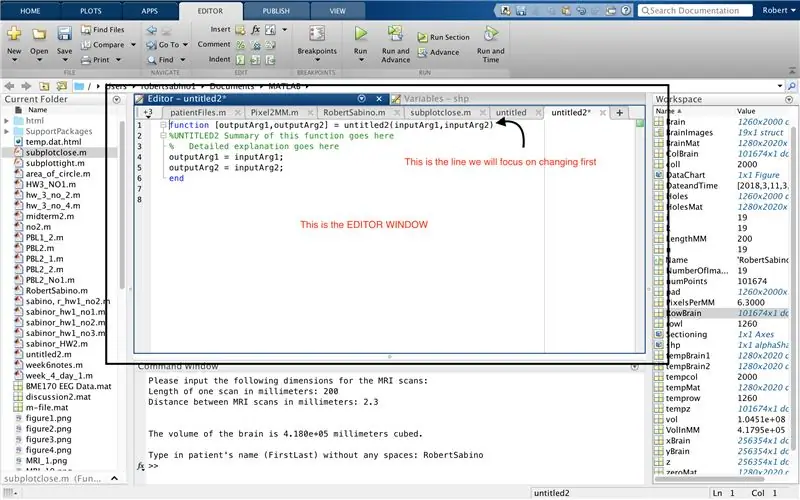

Primer, assegureu-vos que l’ordinador hagi descarregat MATLAB_R2017b (baixeu-lo aquí) i obriu MATLAB. A la interfície MATLAB, feu clic al botó que hi ha a l'extrem superior esquerre de la finestra que diu "Nou" amb un signe més groc en negreta i seleccioneu l'opció "funció" per obrir a la finestra de l'editor un espai que s'assembli a tercera imatge. Ens centrarem en canviar la primera línia per configurar la funció. On es diu "outputArg1", substituïu-lo per "brain", "outputArg2" per dir "forats", "sense títol2" a "exp2" i "inputArg1" a "imatge" i suprimiu "inputArg2". Ara teniu una funció que s'anomena "exp2", prenent un argument "imatge" i sortint els límits de "cervell" i "forats". La primera línia de la funció hauria d’assemblar-se a la línia representada a la quarta imatge. Suprimiu tot el codi que hi ha a sota d'aquesta línia inicial.
Pas 4: Desenvolupament de l'algorisme Bound: Cerca de Bounds

Escriviu el codi de la manera següent a sota de la línia. Aquesta secció de la funció fa el següent línia per línia.
- Carregueu la imatge "imatge" a la variable "mri".
- Convertiu "mri" en una imatge feta de valors en un rang de nombres a uns i zeros (també conegut com a binarització) basat en un valor llindar establert. Si el valor d’un píxel és igual o superior a 0,1, s’estableix a un, si no, el valor d’aquest píxel s’estableix a zero.
- Les quatre línies següents converteixen en zeros 10 columnes i files a les vores de la capa d’exploració de ressonància magnètica, per evitar la lectura de valors inadequats com a límits (tal com s’ha après experimentant amb el codi).
- A la línia final, bwboundaries traça els límits de la imatge binaritzada "mri" i la posa igual a "b", una matriu amb els elements els índexs dels quals corresponen als dels límits establerts a un.
Pas 5: Desenvolupament de l'algorisme Bound: generació de matriu de Bound exterior
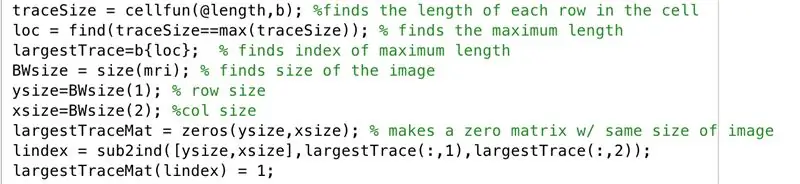
Seguiu a la finestra de l'editor amb el següent codi a la imatge. Aquesta secció del codi fa el següent línia per línia.
- Cerqueu la longitud de cadascuna de les files de la imatge binaritzada "b" (cellfun aplica la funció de longitud a cada fila).
- Establiu "loc" per emmagatzemar les longituds màximes.
- Cerqueu l'índex de la longitud màxima, configurat per emmagatzemar-lo a "més granTrace".
- Cerqueu la mida de la imatge "mri", que consta de la mateixa mida que "b", i configureu-la com a "BWsize".
- Cerqueu el nombre de files de la matriu de la imatge, configurat com a "ysize".
- Cerqueu el nombre de columnes de la matriu de la imatge, configurat com a "xsize".
- Genereu la matriu "largestTraceMat", una matriu de zeros "ysize" per "xsize".
- Cerqueu l'índex equivalent dels valors subscrits corresponents a on estaven els valors més grans de Trace x i els valors y, emmagatzemeu-los al vector "lindex".
- A la matriu de zeros, "largestTraceMat", converteix els elements en els índexs que corresponen als valors d'índex emmagatzemats com a elements a "lindex" en uns.
Per tant, la matriu lògica "largestTraceMat" té la regió delimitada més gran de la secció transversal d'escaneig cerebral donada representada com aquelles amb un teló de fons de zeros
Pas 6: Desenvolupament de l'algorisme Bound: Treballar amb el punt central

A continuació, hem de provar si la secció transversal consta de més d'una regió (la més gran). En provar l'alineació del centre de la regió més gran, podem veure si hi ha una regió contigua, que donaria un centre de centre més centrat, o la possibilitat de múltiples regions.
- Utilitzeu "regionProps" per trobar informació sobre els centreids presents, iguals a la matriu d'estructures "tempStruct"
- Formulari matriu "centroids" amb dades del camp "centroide" concatenat verticalment
- Agafeu els valors de la segona columna de "centroids" (les coordenades de la dimensió horitzontal)
- Executeu un filtre per comprovar l'alineació del centre per al centre horitzontal
Pas 7: Desenvolupament de l'algorisme Bound: quan un Centroid no està centrat
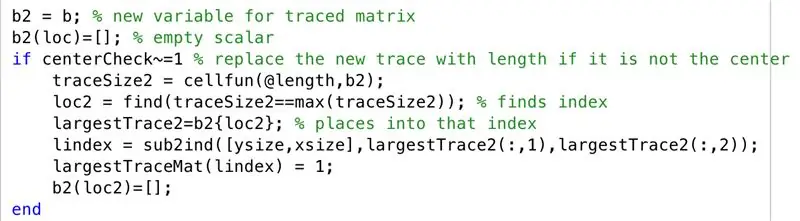
En el cas que el centre de la regió de traça més gran no estigui centrat, passem pels següents passos. Com havíem observat a les ressonàncies de ressonància magnètica, la tendència era que els hemisferis del cervell es retratessin a la secció transversal quan no fossin contigus, de manera que ara continuem traçant la segona traça més gran junt amb la traça més gran de "largestTraceMat"
- Estableix la matriu traçada en una nova variable "b2"
- Inicialitzeu la matriu buida "b2", amb un conjunt indexat per "loc"
- Creeu un condicional per a quan un centre no estigui centrat (és a dir, una capa de diverses regions)
- Definiu una nova mida de traça per a cada fila (traceSize2)
- Establiu "loc2" per trobar els índexs on hi ha límits
- Deixeu que les cel·les especificades per "loc2" a "b2" siguin iguals a "trace2 més gran"
- Convertiu els índexs en índexs, definiu-los com a "lindex"
- Canvieu els elements corresponents a "lindex" a "largestTraceMat" a 1
- Inicialitzeu la matriu buida "b2", amb un conjunt indexat per "loc2"
Pas 8: Desenvolupament de l'algorisme Bound: interferència de forats

Quan es tracta de forats, els valors emmagatzemats a "b2" mantenen un seguiment d'estructures diferents de la traça més gran, i traçar-les en una forma plena de "TraceMat més gran" revelarà on hi ha forats a les regions del cervell.
- Creeu una matriu "cheaMat", que és un formulari completat de "més granTraceMat"
- Creeu una matriu "interferenceMat", una matriu de zeros "ysize" per "xsize"
- Creeu una matriu "interferenceloc", per emmagatzemar els valors de "b2", concatenats verticalment
- Creeu una matriu "lindex" per emmagatzemar els índexs que corresponen a "interferenceloc"
- Per als índexs de "interferènciaMat" que corresponguin a "lindex", establiu el valor a 1, fent una regió delimitada diferent
Pas 9: Desenvolupament de l'algorisme Bound: localització de forats, finalització de límits cerebrals i forats

- Estableix la matriu "tempMat" igual a "interferencyMat" més "fillingMat", afegint així cada valor de la matriu entre si
- Estableix la matriu "holesLoc" igual als índexs en què "interferènciaMat" i "omplertMat" eren iguals a un
- Configureu "holesMat" com a matriu zero de dimensions "ysize" x "xsize"
- Establiu índexs a "holesMat" que siguin iguals a "holesLoc" que els altres
- Estableix "cervell" a "més granTraceMat"
- Estableix "orificis" a "orificisMat"
Amb la troballa on els valors de les matrius afegides eren iguals a 2, les ubicacions dels forats es van assegurar fàcilment i es van representar en una matriu buida.
Pas 10: registre de dades: funció PatientFiles.m
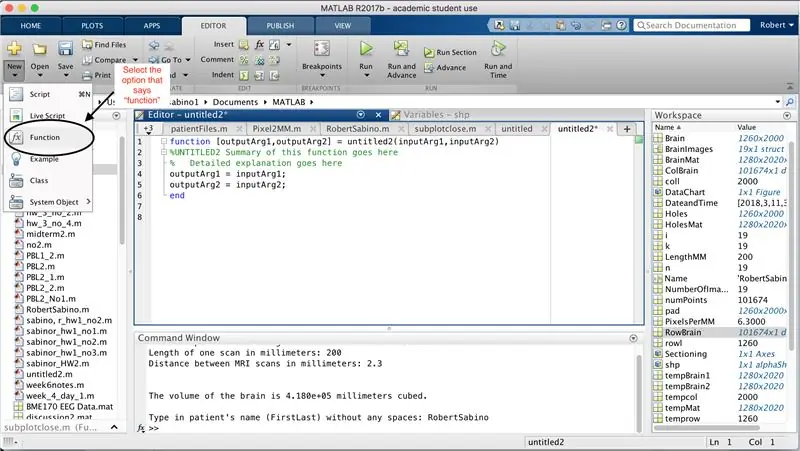
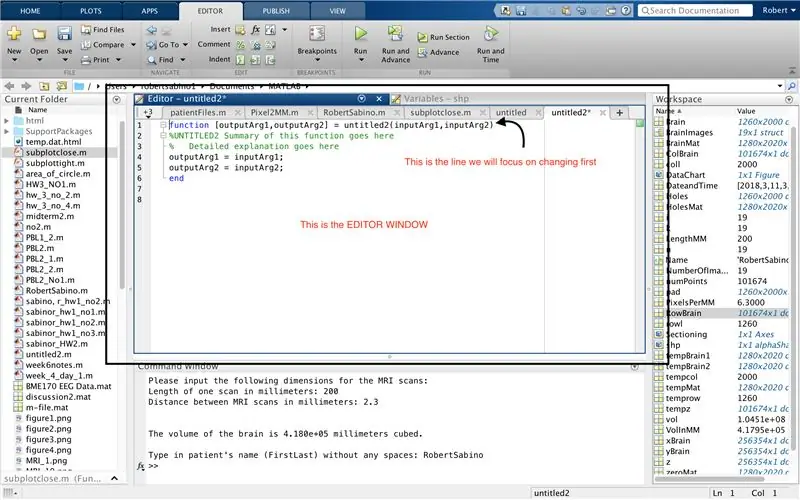

De la mateixa manera que la configuració de l'última funció, feu clic al botó de l'extrem superior esquerre de la finestra que diu "Nou" amb un signe més groc en negreta i seleccioneu l'opció "funció" per obrir a la finestra de l'editor un espai que s'assembla a la de la tercera imatge. A la primera línia, suprimiu la matriu de sortida i substituïu-la per simplement "sortida", substituïu "untitled2" per "fitxers pacient", suprimiu tots els arguments d'entrada i, en canvi, seguiu el format especificat a la quarta imatge de la línia de codi. La primera línia d'aquesta funció hauria de coincidir amb el format de la imatge.
Pas 11: registre de dades als fitxers
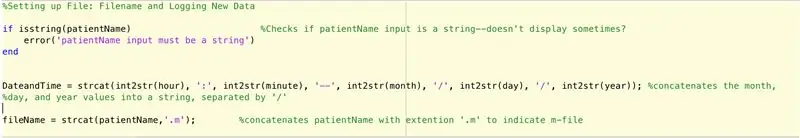

Per configurar un fitxer per registrar les dades trobades per la funció principal (encara per descriure), hem de seguir aquests passos (tal com prescriu el codi línia per línia).
- Comproveu si l'entrada de pacientName és una cadena.
- Si no és una cadena, mostreu que l’entrada pacientName hauria de ser una cadena.
- Finalitzeu la sentència if (eviteu l'error).
- Configureu una declaració de cadena "DateandTime" que donarà el format següent: hora: minuts - mes / dia / any.
- Estableix la variable fileName al següent: nom_pacient.m.
Ara a la següent secció de la funció: Ja existeix un fitxer d’aquest nom?
1) Suposem que el fitxer d’aquest nom ja existeix:
- Executeu el fitxer per obtenir els valors del passat a la cua
- Afegiu les dades "DateandTime" de la iteració actual com a cel·la nova a la matriu de cel·les de valors x (final d'índex + 1)
- Afegiu el valor actual "brainVolume" com a cel·la nova a la matriu de cel·les de valors y (final d'índex + 1)
- Deseu les variables actuals carregades al fitxer.
2) Suposem que el fitxer d’aquest nom no existeix:
- Creeu un fitxer nou amb el nom emmagatzemat a la variable "pacientName"
- Afegiu les dades actuals "DateandTime" com a cel·la a la matriu de cel·les buida de x valors
- Afegiu les dades actuals "brainVolume" com a cel·la a la matriu de cel·les buida de valors y
- Deseu les variables actuals carregades al fitxer.
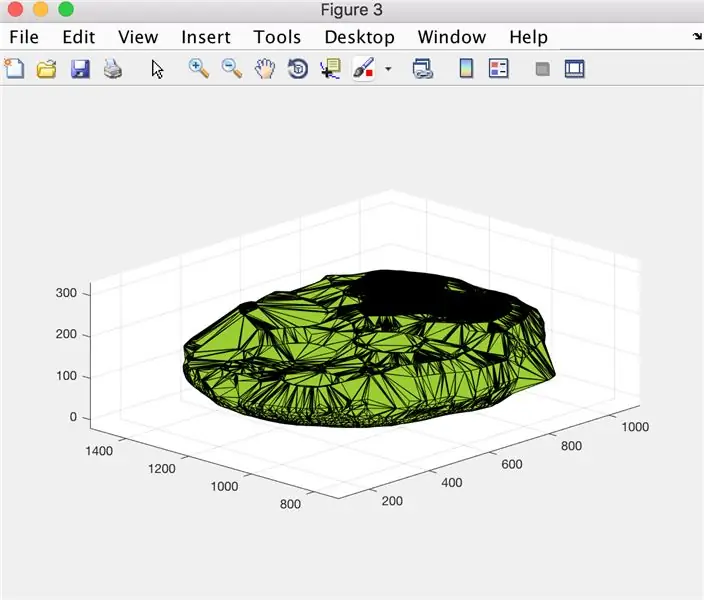
Pas 12: registre de dades: visualització d'una parcel·la de volum cerebral al llarg del temps

- Convertiu la matriu de valors x (xVals) en una matriu categòrica (xValsCategorical), per permetre el traçat
- Genera la finestra de la figura 5
- Representa els punts designats per "xValsCategorical" i "yVals" (que contenen volum cerebral), utilitzant cercles buits per indicar punts i que es connecten mitjançant línies discontínues
- Títol de la trama com: pacientNom Dades de volum cerebral
- Etiqueu l'eix x tal com es mostra a la imatge
- Etiqueu l'eix y tal com es mostra a la imatge
- Sigui la figura 5 igual a la sortida
A partir d’això, la funció pacientName que s’anomena generarà un fitxer amb dades editades que fa un seguiment del volum cerebral al llarg del temps i una trama que mostra les tendències.
Pas 13: Tancament de les llacunes a les subtrames: Subplotclose.m

La funció, adaptada del codi de https://www.briandalessandro.com, funciona per tancar els buits entre les figures de subtrama del codi principal, quan es creen les figures que mostren les imatges de ressonància magnètica i les capes cerebrals. La funció de subtrama utilitzada dins de subtrotclose.m ajusta la posició de les subtrames donades per ajustar-se perfectament entre si en l'aspecte de la dimensió més llarga. Per exemple, si el codi té una matriu de 7 x 3, les files s’adaptaran perfectament ja que la dimensió de la fila és més llarga. Si el codi té una matriu de 3 x 7, les columnes s’adaptaran perfectament, amb buits a les files, tal com es mostra a les figures del nostre codi principal.
Pas 14: el codi principal: esborrar-ho tot i sol·licitar entrades

Per iniciar el codi principal, feu clic al mateix botó que diu "Nou" a l'extrem superior esquerre de la finestra i seleccioneu "Script" en lloc de "Funció" a les seccions anteriors. Escriviu el codi tal com es mostra a la imatge a la finestra de l'editor. Les línies de codi fan les tasques següents en ordre:
- Tanqueu tots els fitxers oberts excepte 0, 1 i 2.
- Tanqueu totes les finestres de les figures.
- Esborreu totes les variables de l'espai de treball.
- Esborreu la finestra d'ordres.
- Visualització a la finestra d'ordres: introduïu les dimensions següents per a les exploracions de ressonància magnètica:
- En una nova línia de la finestra d'ordres, pregunteu: Longitud d'un escaneig en mil·límetres:. La resposta de l'usuari es definirà a la variable "lengthMM".
- En una nova línia, pregunteu: Distància entre exploracions de ressonància magnètica en mil·límetres:. La resposta de l'usuari es definirà a la variable "ZStacks".
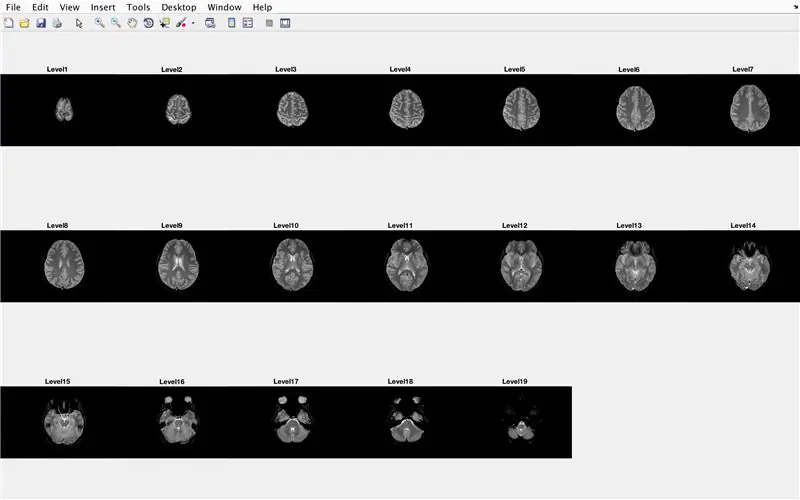
Pas 15: el codi principal: processament per lots de les imatges

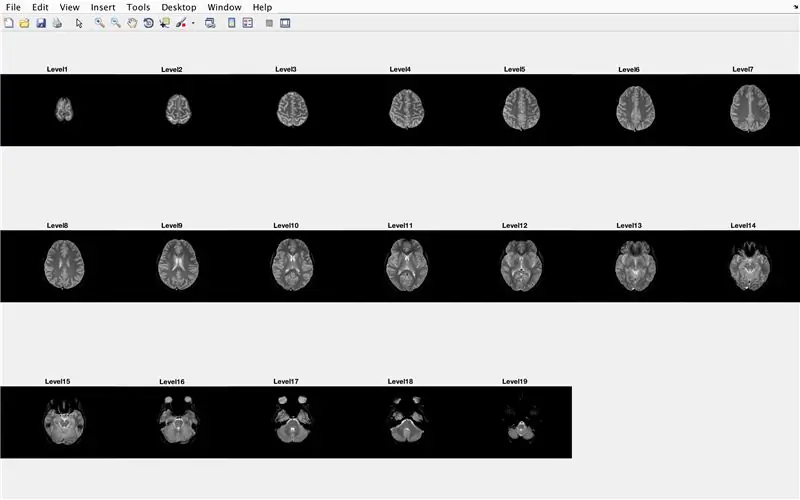
En aquesta secció, el codi carregarà les imatges (que consisteixen en exploracions de ressonància magnètica de les seccions transversals del cervell) i emmagatzemarà els noms de cada fitxer d'imatges a la variable "Base" i mostrarà cadascuna de les exploracions de ressonància magnètica. Seguiu amb el codi de la imatge, que fa el següent:
- Creeu una matriu d'estructures "BrainImages" que contingui informació sobre tots els fitxers de la carpeta actual que s'adaptin al format de nom de MRI _ (). Png
- Estableix la variable "NumberofImages" igual al nombre d'elements de la matriu d'estructures "BrainImages"
- Obriu la finestra de la figura 1
- Establiu un bucle for per fer un recorregut pel nombre d'imatges comptades al fitxer
- Per a cada bucle, "CurrentImage" és el nom respectiu de cada fitxer MRI_i.png, amb el número d'iteració com a "i"
- Genereu la subtrama a 3 x 7 per mostrar les 19 imatges que carregarà "imshow"
- Mostra cada imatge com un element més a la finestra de la figura de la subtrama
- Anomeneu cada element de subtrama com a Nivell_, on en blanc és el número d'iteració del bucle for.
- Finalitzar el bucle for (evitant errors)
Es mostrarà a la finestra de la figura 1 totes les exploracions de ressonància magnètica en format brut en una configuració de 3 x 7 sense buits en l'orientació x.
Pas 16: el codi principal: encoixinat

Amb el farciment, evitem que es produeixin lleugeres discrepàncies en les mides de la imatge que puguin produir un error de desajust de dimensions en el cas que una imatge sigui una mica més gran que una altra.
- Obriu la finestra de la figura 2
- Carregueu la matriu d'imatges de MRI_1-p.webp" />
- Cerqueu la mida de la matriu de la imatge i configureu-lo a "OriginalXPixels" (per al nombre de files) i "OriginalYPixels" (per al nombre de columnes)
- Configureu la matriu "BrainMat" perquè consti de tots els zeros amb 20 files més i 20 columnes més per a cada pla i 19 seccions transversals totals, una per pla.
- Configureu "HolesMat" perquè consti de la mateixa matriu tridimensional de zeros per introduir les coordenades del forat més endavant
- Creeu "zeroMat" per tenir la mida del bloc de notes més vint files i vint columnes, una matriu bidimensional de zeros.
Pas 17: el codi principal: determinar límits
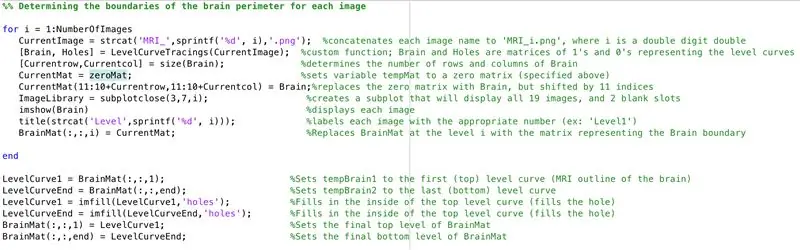
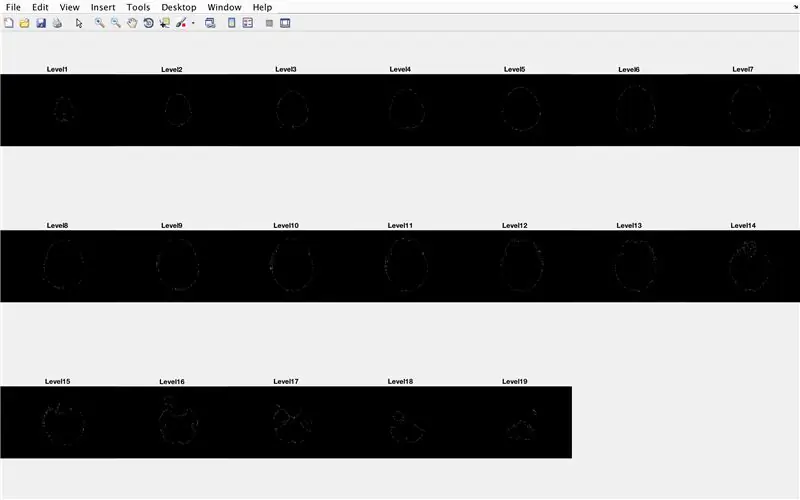
- Establiu un bucle for per examinar les dades de cada imatge carregada anteriorment
- De la mateixa manera que es processava per lots anteriorment, "CurrentImage" carrega els fitxers amb "MRI_i.png", on i és el número d'iteració
- Executeu cada imatge mitjançant la funció de processament "LevelCurveTracings2.m" que heu creat anteriorment
- Cerqueu la mida de la sortida "Cervell", configureu el nombre de files a "Currentrow" i el nombre de columnes a "Currentcolumns".
- Establiu "CurrentMat" a una matriu de zeros amb les dimensions especificades per "Currentrow" i "Currentcolumns"
- Centreu les dades de "Brain" a "CurrentMat", amb un marge de 10 files per tots els costats
- Genereu una subtrama de dimensions 3 x 7 per mostrar els límits de les imatges
- Títol de cadascun dels elements de la subtrama a la finestra de la figura
- Genereu una matriu tridimensional "BrainMat" formada per cada capa de límits "CurrentMat"
- Finalitza el bucle for (per evitar errors)
La subsecció següent omple els forats deixats a la part superior i inferior de la forma tridimensional proposada
- Estableix "LevelCurve1" igual a la primera capa de "BrainMat" (part inferior del sòlid)
- Estableix "LevelCurveEnd" igual a la capa final de "BrainMat" (part superior del sòlid)
- Sobreescriu "LevelCurve1" amb una capa completa
- Sobreescriu "LevelCurveEnd" amb una capa completa
- Estableix la capa completa com a capa inferior de "BrainMat"
- Estableix la capa emplenada com a capa superior de "BrainMat"
Pas 18: el codi principal: determinar la dimensió Z adequada

Les tres primeres línies consisteixen en configurar una matriu buida "z" i fer operacions de conversió senzilles (dividir píxels per longitud) per obtenir una lectura adequada del volum en mm ^ 3
- Creeu un bucle for per recórrer cada capa
- Cerqueu el nombre d’unes en una capa determinada
- Convertiu les coordenades z d'aquestes en valors que s'escalen a la proporció adequada, establerts a "tempz", un vector de columna
- Afegiu el valor z de la corba de nivell al vector z
Amb això, les coordenades z s’ajusten correctament.
Pas 19: el codi principal: determinar les coordenades X i Y

Ara determinem les posicions x i y de cadascun dels punts dels límits.
- Inicialitzeu "xBrain" com a matriu buida
- Inicialitzeu "yBrain" com a matriu buida
- Configureu un bucle for per repassar cada imatge carregada
- Compileu una matriu de dues columnes per emmagatzemar les coordenades planes de cada punt al límit, representades pels vectors de columna "RowBrain" i "ColumnBrain"
- Afegiu "xBrain" amb les coordenades "RowBrain" que es troben actualment
- Afegiu "yBrain" amb les coordenades "ColumnBrain" que es troben actualment
- Finalitzar el bucle for (per evitar errors)
Pas 20: el codi principal: traçar una estructura tridimensional, trobar volum i registrar dades
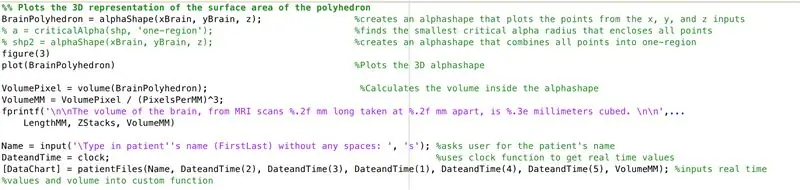

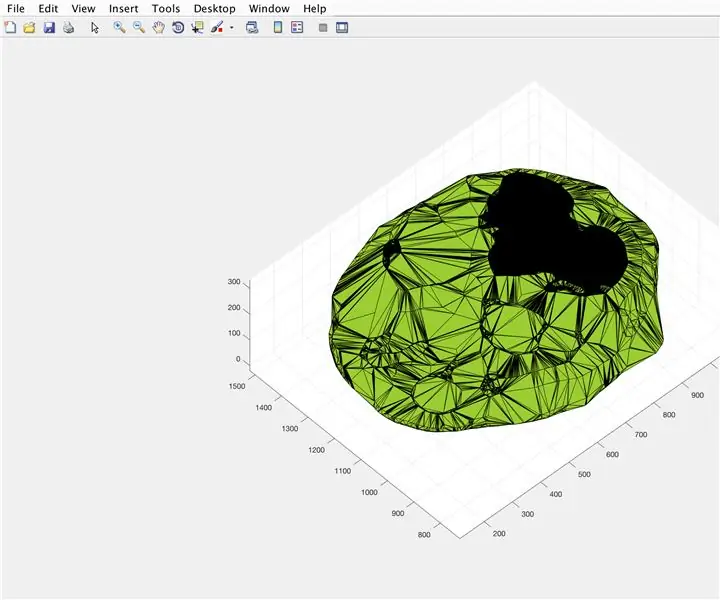
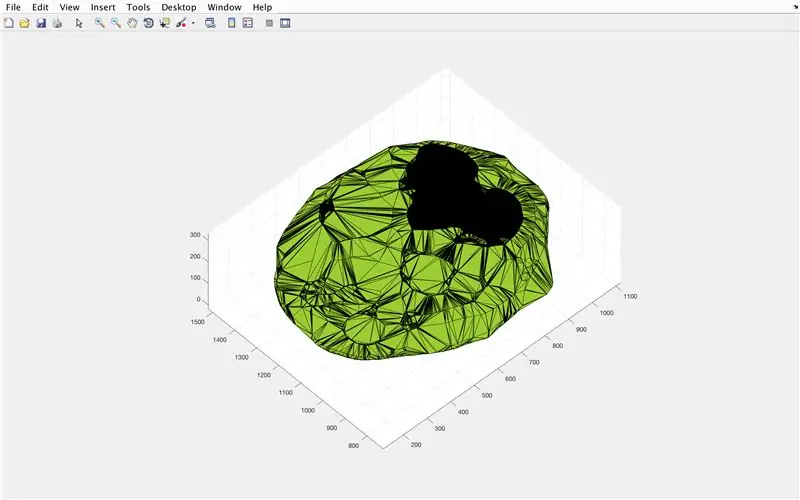
Mitjançant la funció alphaShape, crearem una estructura tridimensional a partir de la qual podem calcular el volum del cervell.
- Utilitzeu la funció alphaShape, connecteu els vectors "xBrain", "yBrain" i "z" per a les coordenades x, y i z, i establiu igual a "BrainPolyhedron"
- Obriu la finestra de la figura 3
- Representa la forma alfa calculada "BrainPolyhedron", que es mostra a la finestra de la figura
- Calculeu el volum de la forma alfa mitjançant una funció "volum" que funciona per a les formes alfa
- Convertiu el volum en mm ^ 3
- Imprimiu el volum del sòlid a la finestra d'ordres
- Sol·liciteu que es defineixi un nom de pacient com a entrada
- Obteniu la data i l'hora actuals amb el rellotge i configureu-les a "Data i hora"
- Truqueu a la funció "patientFiles" per registrar i representar les dades calculades
A partir d'aquí, la segona i la tercera imatge haurien de mostrar les figures que apareixen i la quarta imatge la que s'hauria de mostrar a la finestra d'ordres.
Recomanat:
Seguiment i seguiment de botigues petites: 9 passos (amb imatges)

Track & trace per a botigues petites: es tracta d’un sistema dissenyat per a petites botigues que se suposa que es munta en bicicletes elèctriques o patinets electrònics per a lliuraments a curt abast, per exemple, una fleca que vulgui lliurar pastes. Què significa Track and Trace? Track and trace és un sistema utilitzat per ca
Kits de cotxes de seguiment de robots intel·ligents de bricolatge Seguiment fotosensible del cotxe: 7 passos

Kits de cotxes de seguiment de robots intel·ligents de bricolatge Seguiment del cotxe fotosensible: dissenyat per SINONING ROBOT Podeu comprar amb el robot de seguiment El xip LM393 compara els dos fotoresistors, quan hi ha un LED de fotoresistència lateral en BLANC, el costat del motor s'aturarà immediatament, l'altre costat del motor girar, de manera que
Time Cube: gadget de seguiment del temps Arduino: 6 passos

Time Cube: gadget de seguiment del temps Arduino: m'agradaria proposar-vos un projecte d'arduino senzill però realment útil per fer un seguiment dels esdeveniments temporals invertint algun gadget de cub intel·ligent. Gireu-lo a "Treball" > " Aprèn " > " Tasques " > " Descansa " costat i comptarà el
Seguiment GPS en temps real: 3 passos
Seguiment GPS en temps real: en aquest tutorial, us mostrarem com fer un seguiment de la ubicació en temps real mitjançant un BerryGPS-GSM, un Raspberry Pi Zero i un estat inicial. Enviarem longitud, latitud i velocitat via 3G amb el BerryGPS-GSM a l’estat inicial
Visualització de temps i temps Arduino 3 en 1: 11 passos

Pantalla de temps i temps Arduino 3-en-1: m'agraden els microcontroladors PIC i m'agrada programar en llenguatge assemblador. De fet, durant els darrers dos anys, he publicat al meu lloc web uns 40 projectes basats en aquesta combinació. Fa poc, estava demanant algunes peces a una de les meves versions preferides dels EUA
